Résumé de l’étude:
Le diagnostic était établi à partir d’un échantillon d’ADN foetal libre prélevé dans le sang maternel, en utilisant l’analyse des polymorphismes du nucléotide simple (SNP ou single-nucleotide polymorphisms en anglais).
Des échantillons de sang ont été prélevés sur 21 femmes adultes enceintes (entre 6 et 21 semaines de gestation) ainsi qu’un échantillon de matériel génétique des pères biologiques correspondants. Des tests de paternité ont été effectués à partir de chaque échantillon sanguin maternel contenant suffisamment d’ADN fœtal libre.
L’ADN de l’enfant a été comparé à celui de son père confirmé ainsi qu’à celui de l’ADN de 1,820 mâles non apparentés et choisis au hasard.
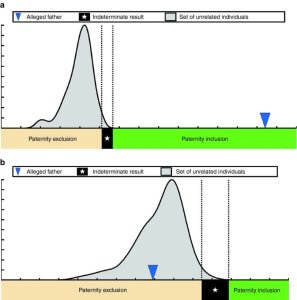
Sur l’image ci-contre, le flèche bleue indique les résultats du test de paternité prénatal. Ainsi, le père présumé est bien le père biologique sur la figure a. Et il ne l’est pas en figure b.
L’un des 21 échantillons de sang maternel ne contenait pas suffisamment d’ADN fœtal libre pour effectuer les 1,821 tests de paternité nécessaires à l’étude. Pour les 20 autres échantillons, les résultats des 20 x 1,821 tests = 36,420 tests sont les suivants :
| Groupes de tests | Père biologique ou pas ? | Résultat |
| 20 tests de paternité sur 20 (100%) | Effectués avec leurs pères biologiques prouvés | Paternité confirmée correctement à 100%. |
| 36,382 tests de paternité sur 36,400 | Effectués avec des mâles non apparentés | Paternité correctement exclue à 99,95%. |
| 18 tests de paternité sur 36,400 | Effectués avec des mâles non apparentés | L’analyse n’a pas été conclusive sur 0,05% des cas. |
Conclusion : 36,400 tests de paternité réalisés sans erreur de diagnostic
Il n’y a pas eu de diagnostic erroné sur les 36,420 tests de paternité effectués, seuls 18 tests n’ont pas permis de conclure sur une confirmation ou exclusion de paternité. Le test de paternité prénatal non invasif basé sur l’analyse des polymorphismes nucléotidiques (SNP) a donc permis de déterminer avec exactitude la paternité en début de grossesse. Des résultats précis ont ainsi été obtenus par cette méthode malgré une faible fraction d’ADN fœtal libre et ce, dès la 6e semaine de grossesse.
Pour en savoir plus, vous trouverez ci-dessous le détail du contexte, de la méthode et la discussion autour de cette étude.
Contexte de l’étude :
Jusqu’à récemment, un test de paternité prénatal fiable nécessitait le prélèvement de villosités choriales ou de liquide amniotique par amniocentèse, ces deux techniques comportant des risques à la fois pour la mère et pour le fœtus. L’amniocentèse comporte un risque de fausse couche compris entre 1/300 et 1/500, tandis que le prélèvement de villosités choriales entraîne un risque similaire de fausse couche ainsi qu’un risque de 1/3000 de dommage d’un membre du fœtus, surtout lorsqu’il est effectué avant la 10e semaine de grossesse. Bien que la Société Américaine des Obstétriciens et Gynécologues recommande que ces interventions invasives soient proposées à toutes les femmes enceintes, elles sont le plus souvent seulement proposées aux mères comportant un risque élevé d’anomalies génétiques, notamment après le test du premier trimestre.
La découverte de l’ADN fœtal libre dans le sang périphérique maternel conduisit les auteurs de l’étude à penser qu’il serait possible de développer un test de paternité prénatal non invasif, qui permettrait d’éviter les risques associés au procédures invasives. Bien que l’ADN fœtal libre ait été connu depuis plus d’une dizaine d’années, il a été très difficile de développer un test de paternité prénatal non invasif fiable utilisant des échantillons de sang maternel périphérique. Il faut savoir que l’ADN libre fœtal représente à peine 20% de l’ADN libre que l’on trouve dans le sang maternel et qu’il est très fragmenté. Les difficultés techniques portaient sur la quantité réduite d’ADN fœtal libre disponible pour l’étude ainsi que sa grande dilution dans le sang maternel. Cette difficulté a été surmontée grâce à la combinaison de l’étude
- des micro réseaux ou du séquençage à très haut débit (elle permet de mesurer des centaines de milliers voir de millions de fragments d’ADN), avec
- des techniques bioinformatiques sophistiquées permettant d’extraire un maximum d’information de l’ADN fœtal libre présent en si petite quantité.
Les auteurs de l’étude avaient publié précédemment une méthode dite « Parental Support », qui utilisait l’analyse des polymorphismes nucléotidiques (en anglais single-nucleotide polymorphism (SNP) microarray measurements) issus de l’ADN maternel et paternel pour améliorer la précision du génotype de l’embryon. Bien que cette méthode permette d’identifier avec précision la ploïdie sur les 24 chromosomes à partir d’une unique cellule embryogénique, cette méthode n’avait pas été employée pour développer un test de paternité.
L’étude actuelle utilise une version de l’algorithme de « Parental Support » pour déterminer si les polymorphismes nucléotidiques (SNP) du fœtus mesurés à) partir du sang maternel pourraient être attribués au père supposé.
Méthode :
Des échantillons de sang ont été prélevés sur 21 femmes adultes enceintes (entre 6 et 21 semaines) ainsi qu’un échantillon de matériel génétique des pères biologiques correspondants. La paternité a été confirmée par le test des enfants à la naissance, des produits de la conception, par le strict contrôle de la fécondation, et/ou le diagnostic génétique de pré-implantation durant la fécondation in vitro. Chaque participant avait donné son consentement éclairé pour participer à cette étude.
Les échantillons d’ADN parentaux ainsi que l’ADN fœtal libre ont été amplifiés par PCR et les polymorphismes nucléotidiques furent analysés informatiquement selon la méthode « HumanCytoSNP-12 array », permettant ainsi que confirmer ou d’exclure la paternité. Des tests de paternité ont été effectués à partir de chaque échantillon sanguin maternel contenant suffisamment d’ADN fœtal libre. L’ADN de l’enfant a été comparé à celui de son père confirmé ainsi qu’à celui de l’ADN de 1,820 mâles non apparentés et choisis au hasard. Il a été vérifié que les personnes mâles non apparentées étaient issues de groupes ethniques suffisamment variés.
Résultats de l’étude :
L’un des 21 échantillons de sang maternel ne contenait pas suffisamment d’ADN fœtal libre pour effectuer les 1,821 tests de paternité nécessaires à l’étude.
- Le test de paternité prénatal a permis de confirmer correctement avec 100% de précision la paternité des 20 échantillons utilisables, avec des valeurs de probabilité inférieures à 0,0001 (une valeur P <10−4 ). Le test de paternité prénatal basé sur l’ADN fœtal libre a permis de retrouver le père biologique prouvé de chacun des enfants, dès la 6e semaine de grossesse même avec une faible fraction d’ADN fœtal libre.
- Pour les 36,400 tests de paternité effectués avec l’ADN de mâles non apparentés, 36.382 tests ont correctement exclu la paternité (99,95%) alors que les résultats des 18 tests de paternité restants étaient indéterminés (ils ne permettaient pas de conclure sur une inclusion ou une exclusion de paternité). Il n’y a pas eu d’erreur de diagnostic sur la paternité parmi les 36,400 tests effectués.
Discussion
Les auteurs de l’étude présentent leur méthode comme la première méthode permettant de tester cliniquement la paternité de manière non invasive avec une telle précision. Les méthodes traditionnelles de tests de paternité post natales portent sur l’analyse de séquences de STR et leur comparaison avec un ou plusieurs pères présumés. Dans la mesure où après la naissance on peut disposer de grandes quantités d’ADN (ne serait-ce que par prélèvement buccal), les mesures sont fiables et seuls 15-20 marqueurs (ou séquences de STR) sont nécessaires pour obtenir des résultats très précis aux tests de paternité postnataux.
Les options sont plus limitées pour déterminer la paternité prénatalement sans avoir recours à des techniques invasives telles que l’amniocentèse ou le prélèvement de villosités choriales, ou lorsque l’on se situe très tôt dans la grossesse. Bien que l’ADN fœtal libre extrait du sang périphérique maternel puisse être mesuré avec fiabilité, et que de nombreuses équipes aient fait état de l’amplification et la détection d’allèles spécifiques du fœtus en utilisant la réaction PCR, ces méthodes ne portaient pas sur une quantité suffisante d’allèles du fœtus pour pouvoir tester la paternité assez précisément et ne se prêtaient pas à l’analyse d’un nombre supérieur de loci.
L’utilisation de l’ADN libre fœtal pour confirmer correctement la paternité a été rapportée récemment dans 30 cas (N Engl J Med 2012;366:1743–1745). Cependant, cette étude comparait seulement le père connu avec un autre mâle non apparenté. Dans le cas où le père biologique ne serait aucun des deux pères testés, cette méthode pourrait entraîner un résultat de paternité chez un individu qui serait simplement plus étroitement lié que l’autre à l’enfant en raison de relations génétiques proches, telles que l’appartenance à de petites sous-populations ethniques. D’où l’importance de tester la paternité contre un grand nombre de mâles non apparentés. Dans cette étude, les tests de paternité avaient été effectués au total sur sur 5,000 individus choisis au hasard.
La méthode ici utilisée permet d’éviter les problèmes liés aux méthodes d’analyse traditionnelles de l’ADN et de l’ADN libre en utilisant l’analyse des polymorphismes nucléotidiques (SNP arrays) à haut débit. Une large quantité d’ADN maternel libre étant présente dans l’échantillon sanguin, ce qui génère généralement un plus faible rapport signal/bruit pour la détermination du génotype fœtal à partir de l’ADN libre fœtal. En identifiant le génotype maternel et la fraction fœtale a permis de prédire la quantité de signal maternel et d’ajuster les réglages en fonction. Tandis que chaque mesure de SNP est le bruit, la compilation de données sur plus de 300,000 SNPs a permis de déterminer ou d’exclure la paternité. Les résultats étaient précis avec des fractions aussi faibles que 2,3% d’ADN libre fœtal / maternel, suggérant qu’il est possible de faire ce test de paternité prénatal au tout début de la grossesse, vers 6 semaines, alors que les fractions fœtales sont encore très faibles.
En résumé, cette étude rapporte la précision des résultats du test de paternité prénatal non invasif qui est actuellement cliniquement disponible (aux Etats-Unis). Les auteurs estiment que « Il conviendrait de procéder à des études complémentaires à plus large échelle pour déterminer précisément à partir du quel nombre minimum de semaines de grossesse la paternité pourrait être confirmée avec le même degré de précision et en confirmer l’efficacité en testant la paternité sur un groupe plus important de participants.
Cliquez ici pour demander un test de paternité prénatal non invasif
Pour en savoir plus:
- Nos sources: voir le texte intégral de l’étude Genet Med 2013:15(6):473–477
- L’enfant est né, il me faut un test de paternité avec échantillon buccal
- Une nouvelle étude prouve la meilleure détection de la trisomie 21 foetale par le test de l’ADN libre foetal
- Notre article complet sur le test de paternité prénatal non invasif
- Le test génétique non invasif de la trisomie 13, 18 et 21 fœtale
- Une mutation génétique fausserait-elle les résultats d’un test ADN?
- Pourquoi fait-on participer les mères aux tests de paternité ?
